细胞平台
精华!一文看懂DNA碱基编辑器发展脉络
2018年10月11日/生物谷BIOON/---基因组编辑技术CRISPR/Cas9被《科学》杂志列为2013年年度十大科技进展之一,受到人们的高度重视。CRISPR是规律间隔性成簇短回文重复序列的简称,Cas是CRISPR相关蛋白的简称。CRISPR/Cas最初是在细菌体内发现的,是细菌用来识别和摧毁抗噬菌体和其他病原体入侵的防御系统。(CAS蛋白基因)
在CRISPR/Cas9系统中,酶Cas9在DNA靶位点上进行切割,其中这种靶位点是这样确定的:一种被称作CRISPR RNA(crRNA)的RNA分子利用它的一部分序列与另一种被称作tracrRNA的RNA分子通过碱基配对结合在一起,形成嵌合RNA(tracrRNA/crRNA),然后,借助crRNA的另一部分序列与靶DNA位点进行碱基配对,以这种方式,这种嵌合RNA就能够引导Cas9结合到这个靶位点上并进行切割。在实际应用时,人们可以将tracrRNA和crRNA作为两种向导RNA(gRNA)或者融合在一起形成单向导RNA(single guide RNA, sgRNA),并被用来引导酶Cas9结合到靶DNA序列上并进行切割,其中Cas9与sgRNA一起被称作Cas9-sgRNA系统。
进一步的研究还证实,CRISPR/Cas9的基因组编辑能力只有在被称作前间隔序列邻近基序(protospacer adjacent motif, PAM)的短片段DNA序列的存在下才成为可能。只有DNA靶位点附近存在PAM时,Cas9才能进行准确切割。再者,PAM的存在也是激活酶Cas9所必需的。
2015年1月,Jesse G. Zalatan等人[1]对sgRNA进行扩展,将sgRNA与模块化的招募RNA结合蛋白的RNA结构域融合在一起而将sgRNA转化为支架RNA(scaffold RNA, scRNA),从而在物理上将DNA结合与蛋白招募活性偶联在一起。
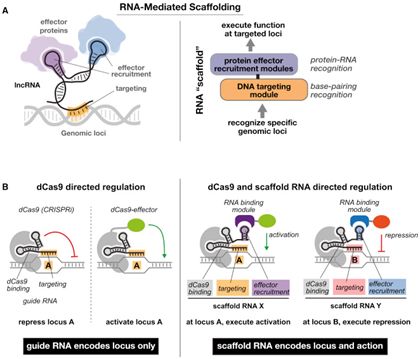
图片来自Cell, 15 January 2015, 160(1):339-350, doi:10.1016/j.cell.2014.11.052。
在广泛使用的CRISPR/Cas9基因编辑工具中,核酸酶Cas9含有两个具有切割活性的结构域:HNH结构域和RuvC结构域,其中HNH结构域切割与crRNA互补的DNA链,而RuvC结构域切割非互补链。RuvC结构域可分为三个亚结构域:RuvC I、RuvC II和RuvC III,RuvC I接近于Cas9的氨基端,RuvC II和RuvC III位于HNH结构域的两侧。仅对Cas9中的RuvC I进行突变,具体而言就是让RuvC I的两个关键氨基酸残基中的一个转换成丙氨酸(D10A或H840A),从而得到Cas9切口酶(Cas9 nickase, Cas9n)。这种切口酶不能切割非互补DNA链,仅能切割与crRNA互补的DNA链。如果同时让Cas9中的这两个结构域发生突变,便可得到仅对DNA有结合活性但没有切割活性的dCas9(nuclease-dead Cas9,没有核酸酶切割活性的Cas9)。
当我们谈论CRISPR/Cas9的应用时,总会出现一个负面因素:同源介导修复(homology-directed repair, HDR)的低编辑效率。与非同源末端连接(non-homologous End Joining, NHEJ)相比,HDR以相对低的频率发生,并且在非分裂细胞中,这个修复机制会进一步遭受下调。
哈佛大学化学家David Liu的实验室不是试图改进HDR的编辑效率,而是构建出新的起着碱基编辑器作用的Cas9融合蛋白。这些融合蛋白含有dCas9或Cas9n和胞苷脱氨酶,它们能够在不切割DNA的情形下将胞嘧啶转化为尿嘧啶,随后通过DNA复制或修复,将尿嘧啶转化为胸腺嘧啶。后来的工作改进了碱基编辑器的灵活性和特异性。新的腺嘌呤碱基编辑器(adenine base editor)还允许人们将腺嘌呤变为肌苷,然后肌苷会转化为鸟嘌呤。
dCas9/Cas9n碱基编辑
构建单碱基编辑器的初始需求:(1)Cas9与胞苷脱氨酶融合在一起;(2)gRNA将Cas9靶向特定的基因组位点;(3)在非靶DNA链的位点4~8上存在胞嘧啶。
2016年,Komor等人[1]利用16个碱基长的XTEN接头(XTEN linker)将大鼠胞苷脱氨酶APOBEC1与dCas9连接在一起,从而构建出第一代碱基编辑器(BE1)。BE1表现出大约5个核苷酸的活性窗口(activity window):靶位点4~8。为了增加体内编辑效率,第二代碱基编辑器(BE2)系统除了将胞苷脱氨酶与dCas9连接在一起之外,还将碱基切除修复抑制剂UGI与dCas9融合在一起,从而将编辑效率提高三倍,最高达到20%左右。对BE1和BE2而言,碱基插入或删除(insertion or deletion, indel)发生率是非常低的(<0.1%),这是因为它们并不直接切割DNA。
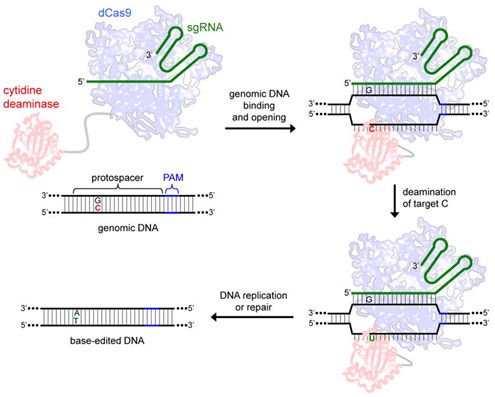
图片来自Nature, doi:10.1038/nature17946。
为了将碱基编辑效率提高到50%以上,人们需要采取一种方法将靶DNA链上的碱基编辑复制到另一条DNA链上。为了做到这一点,Komor等人将dCas9换为Cas9n来模拟错配修复,从而构建出第三代碱基编辑器(BE3)。BE3在非互补DNA链上产生切口,使得它在细胞看来是“新合成的”。因此,细胞使用含尿嘧啶(U)的DNA链作为模板来进行修复,从而复制这种碱基编辑。
在人细胞系的多种靶基因中,这种BE3系统将碱基编辑效率增加到30%以上,它的平均indel发生率仅为1.1%。对这些测试的靶基因而言,这些数字是对Cas9介导的HDR的巨大改进;平均的HDR介导的编辑频率仅为0.5%,并且相比于之前的单碱基编辑,更多的indel被观察到。在多次细胞分裂中,CRISPR碱基编辑持续存在,这表明这种方法产生稳定的碱基编辑。然而,这种BE3系统也会遭受基于Cas9而不是APOBEC1的脱靶活性产生的脱靶编辑。
让碱基编辑变得更好
2016年,Nishida等人[2]在一项研究中通过展示碱基编辑变异体的益处而进一步证实了碱基编辑的实用性。他们通过将来自海鳗(sea lamprey)的胞苷脱氨酶AID(activation-induced cytidine deaminase, 活化诱导的胞苷脱氨酶)与Cas9n融合在一起而构建出Cas9n-AID碱基编辑器。2017年,Komor等人[3]也利用其它的脱氨酶--- AID、CDA1和APOBEC3G---构建出BE3变异体。CDA1-BE3和AID-BE3要比BE3对碱基G之后的碱基C更加高效地编辑,但是APOBEC3G表现出难以预测的序列偏好性。这些结果证实这种特定的脱氨酶和将这种脱氨酶附着到Cas9上的方法影响碱基编辑作用和效率。
2017年,Kim等人利用天然的和经过基因改造的Cas9变异体开发出5种具有不同PAM序列的新型碱基编辑器,从而扩大可进行碱基编辑的靶位点数量。对每种碱基编辑器而言,他们观察到编辑活性最小为50%左右,并且证实每种融合蛋白保持Cas9蛋白在未融合时的PAM性质。Kim等人也让每种碱基编辑器中的胞苷脱氨酶部分发生突变,从而构建出编辑窗口小到1~2个核苷酸的SpCas9碱基编辑器。
为了减少与碱基编辑相关的脱靶效应,Rees等人构建出HF-BE3,即一种含有高保真度Cas9变异体HF-Cas9的碱基编辑器。他们随后利用具有较高的脱靶切割率的gRNA测试了原始的BE3和HF-BE3,结果发现HF-BE3的脱靶编辑比BE3少了37倍,仅在靶编辑效率略有下降。为了进一步提高特异性,他们纯化了HF-BE3蛋白,用于将核糖核蛋白颗粒(RNP)递送到斑马鱼胚胎和小鼠内耳中。这些激动人心的研究进一步展示了碱基编辑有潜力精确修饰基因组。
第四代碱基编辑器
第三代碱基编辑能够产生复杂的产物;人们已观察到不想要的C->G或C->A转换,而不是始终如一的C-> T转换。2017年,Komor等人猜测这些副产物是在碱基切除修复期间尿嘧啶N-糖基化酶(UNG)切除引起的。虽然第三代碱基编辑器包含UNG抑制剂UGI,但是添加第二个UGI拷贝可提高碱基编辑产物的纯度。此外,Komor等人通过延长APOBEC1-Cas9n和Cas9n-UGI中的接头长度提高了碱基编辑产物的纯度,这三项改进一起代表了第四代碱基编辑器(BE4)。与BE3相比,BE4使得C->G和C->A基因编辑产物减少了2.3倍,并且让indel发生率减少了2.3倍。
为了进一步降低第四代碱基编辑器中已经很低的indel发生率,Komor等人将噬菌体蛋白Gam融合到BE4的氨基端上。Gam结合到DNA双链断裂(DSB)的自由端,这可能导致细胞死亡而不是NHEJ修复,因而这会清除碱基编辑细胞群体中的这些发生DNA双链断裂的细胞。将Gam添加到BE4或SaBE4中可将indel发生率降低1.5~2倍。
在很短的时间内,科学家们已将碱基编辑领域扩展到BE2和BE3之外。各种Cas9变体、胞苷脱氨酶和辅助蛋白的使用已构建出一套具有互补和重叠功能的碱基编辑器。碱基编辑的实用性在不断提高。
腺嘌呤碱基编辑器
直到最近,碱基编辑仅限于靶DNA链上的C->T转换,或互补DNA链上的G->A转换。Gaudelli等人寻求构建腺嘌呤碱基编辑器(adenine base editor, ABE),它将腺嘌呤转化为肌苷,从而导致A->G变化。鉴于没有已知的DNA腺嘌呤脱氨酶,他们使用定向进化从RNA腺嘌呤脱氨酶TadA中产生一种。
经过七轮分子进化,Gaudelli等人[7]获得了四种腺嘌呤碱基编辑器:ABE7.10、ABE 6.3、ABE 7.8和ABE 7.9。ABE7.10是最活跃的编辑器,它表现出53%的平均编辑效率,它的编辑窗口为靶位点4~7。ABE 6.3、ABE 7.8和ABE 7.9具有略宽的编辑窗口:靶位点4~9,不过在靶位点8~9上的编辑效率可能更低。尽管胞嘧啶碱基编辑器经常产生混合的碱基编辑细胞群体,但是ABE在靶位点上并没有表现出显著的A到非G的转换。Gaudelli等人提出从DNA中移除肌苷可能并不常见,从而阻止诱导碱基切除修复。
在脱靶效应方面,ABE也比其他的方法更有优势。在与Cas9的面对面的直接比较中,Cas9修饰了12个已知的脱靶位点中的9个,具有14%的indel发生率,而ABE7.10仅修改了12个已知的脱靶位点中的4个,具有1.2%的indel发生率。尽管Gaudelli等人没有对ABE的特异性开展过完整的全基因组研究,但是他们的其他实验提示着ABE是稳健的但又特异性的编辑器。(生物谷 Bioon.com)
参考资料:
1.Jesse G. Zalatan et al. Engineering Complex Synthetic Transcriptional Programs with CRISPR RNA Scaffolds. Cell, 15 January 2015, 160(1):339-350, doi:10.1016/j.cell.2014.11.052
2.Alexis C. Komor et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature, 19 May 2016, 533(7603), 2016):420-424, doi:10.1038/nature17946
3.Keiji Nishida et al. Targeted nucleotide editing using hybrid prokaryotic and vertebrate adaptive immune systems. Science, 353(6305):aaf8729, doi:10.1126/science.aaf8729
4.Alexis C. Komor et. Improved base excision repair inhibition and bacteriophage Mu Gam protein yields C:G-to-T:A base editors with higher efficiency and product purity. Science Advances, 2017 Aug 30;3(8):eaao4774, doi: 10.1126/sciadv.aao4774
5.Y Bill Kim et al. Increasing the genome-targeting scope and precision of base editing with engineered Cas9-cytidine deaminase fusions. Nature Biotechnology, 2017 Apr;35(4):371-376, doi: 10.1038/nbt.3803
6.Holly A. Rees et al. Improving the DNA specificity and applicability of base editing through protein engineering and protein delivery. Nature Communications, 2017 Jun 6;8:15790, doi:10.1038/ncomms15790
7.Nicole M. Gaudelli et al. Programmable base editing of A?T to G?C in genomic DNA without DNA cleavage. Nature, 23 November 2017, 551(7681):464–471, doi:10.1038/nature24644
(本文来源生物谷)
想要了解更多请关注“中洪博元医学实验帮 ”
上一篇:NEJM:临床试验首次表明联合使用免疫疗法和化疗显著延长三阴性乳腺癌患者的寿...
下一篇:JCI:特殊免疫细胞有望成为治疗三阴性乳腺癌的潜在靶点...
上海展辉生物技术有限公司
地址:上海市浦东新区东方路
联系电话:400-689-6719
联系邮箱:3369352092@qq.com
联系我们:
4006896719

扫一扫
关注东洪博元更多动态